Objetivos
General
-
Utilizar espectrofotometría UV-Vis para obtener una curva de calibración la cual permitirá relacionar la concentración de un analito con la absorbancia obtenida, misma que se empleará más adelante para la obtención de enzimas.
Utilizar espectrofotometría UV-Vis para obtener una curva de calibración la cual permitirá relacionar la concentración de un analito con la absorbancia obtenida, misma que se empleará más adelante para la obtención de enzimas.
Específicos
-
Obtener la curva de calibración para:
-
Azúcares reductores (glucosa) usando el método de DNS (ácido 3,5-dinitrosalicílico)
- Proteínas aplicando el método de Bradford
Obtener la curva de calibración para:
- Azúcares reductores (glucosa) usando el método de DNS (ácido 3,5-dinitrosalicílico)
- Proteínas aplicando el método de Bradford
Introducción
Para el análisis de la concentración de una sustancia en específico se requiere conocer que técnica es utilizada para dicho caso; para esta práctica fue utilizada la espectrofotometría UV-Vis es una técnica analítica que permite determinar la concentración de un compuesto en solución. Su teoría está basada en que las moléculas absorben las radiaciones electromagnéticas y a su vez que la cantidad de luz absorbida depende de forma lineal de la concentración. Para hacer este tipo de medidas se emplea un espectrofotómetro, en el que se puede seleccionar la longitud de onda de la luz adecuada que pasa por una solución (la longitud de onda utilizada debe ser la máxima, ya que se desea conseguir los mejores resultados posibles en un punto tal que la sustancia produzca el máximo de absorbancia) y medir la cantidad de luz absorbida por la misma.
La espectrofotometría UV-Vis utiliza la radiación del espectro electromagnético cuya longitud de onda está comprendida entre los 100 y 800 nm, y su efecto sobre la materia es producir transiciones electrónicas atómicas y/o moleculares de las sustancias. La energía que puede absorber una especie química causa la transición de un electrón de un estado basal o fundamental a uno excitado; esta energía de transición está relacionada con la longitud de onda de la radiación a través de la ecuación de Planck.
La base de la espectrofotometría de absorción UV-Vis está fundamentada en la ley de Lambert-Beer, la cual establece que la absorbancia de una solución es directamente proporcional a la concentración del analito en esa solución. Por ello esta técnica se puede emplear para determinar la concentración de un compuesto en una solución. La transmitancia T y la absorbancia se pueden relacionar de la siguiente manera:
A= - log T
Una forma de representar los resultados obtenidos en una espectrofotometría UV-Vis es la curva de calibración es una representación gráfica de un patrón que se mide en función de la concentración del analito con la absorbancia obtenida; esto quiere decir que se preparan soluciones de diferentes concentraciones de este para obtener el valor de absorbancia para cada una de ellas. Los valores de absorbancia obtenidos se leen en λmax. Al obtener la curva de calibración, puede ser comparada con un curva estándar del analito en base a concentraciones de analito conocidas.
Al momento de graficar los datos es necesario saber que en el eje X va la concentración del analito de interés y en el eje Y se localizan las absorbancias medidas.
El comportamiento habitual que se desea con los puntos obtenidos es el de una recta, la cual recibe el nombre de recta de calibrado, y esta se encuentra definida por una ordenada al origen (b) y una pendiente (m), mediante la ecuación y = mx + b. La confiabilidad y exactitud de la curva se hace uso del método de mínimos cuadrados, utilizado con la finalidad de conseguir que la recta formada al unir los distintos puntos no tenga un valor de R2 < 0.98, ya que siendo así la recta no es considerada confiable, debido a la inexactitud que tiene.
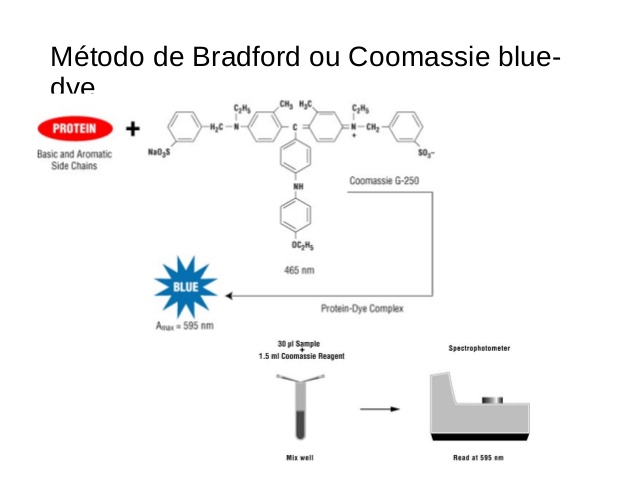
El método de Bradford presenta una relevante importancia, ya que este permite determinar la concentración de proteínas en solución. El principio básico de reacción del colorante azul brillante de coomassie G-250 es el siguiente:
Imagen 1. Mecanismo de la formación del complejo proteína - colorante
Como se puede observar en la imagen 1, la parte aniónica del colorante se une a la proteína, y la unión se distingue a simple vista debido a que la coloración azul que presenta el complejo proteína - colorante. Después de la formación de la formación mencionada anteriormente se prosigue a realizar el análisis de la absorbancia a 595 nm.
Así mismo el método DNS (ácido 3,5 - dinitrosalicílico) es utilizado para determinar los azúcares reductores presentes en una solución. El método se fundamenta principalmente en la reducción del DNS (de color amarillo) por la glucosa u otro azúcar reductor al ácido 3-amino-5-nitrosalicílico (de color rojo ladrillo), cuya presencia puede detectarse por lectura de la absorbancia en la zona de 540-600 nm. Este mecanismo de reacción puede observarse en la imagen siguiente:
Imagen 2. Mecanismo de reacción del método DNS (referencia)
Métodología
Imagen 3. Metodología para cuantificación de azúcares reductores
Imagen 4. Metodología para determinación de proteínas
Resultados
→ Curva tipo de albúmina de huevo (AH)
Tabla 1. Primer curva de calibración
| |
Soluciones
|
Absorbancia
|
1
|
-0.001
|
2
|
0.052
|
3
|
0.088
|
4
|
0.119
|
5
|
0.158
|
6
|
0.198
|
7
|
0.232
|
8
|
0.251
|
9
|
0.340
|
10
|
0.400
|
11
|
0.442
|
Tabla 2. Segunda curva de calibración
| |
Soluciones
|
Absorbancia
|
1
|
-0.245
|
2
|
0.059
|
3
|
0.082
|
4
|
0.122
|
5
|
0.135
|
6
|
0.181
|
7
|
0.216
|
8
|
0.322
|
9
|
0.302
|
10
|
0.362
|
11
|
0.399
|


→ Curva patrón de glucosa
Tabla 3. Primera curva de calibración
Soluciones
Absorbancia
1
-0.001
2
0.001
3
0.073
4
0.195
5
0.366
6
0.534
7
0.685
8
0.834
9
1.032
10
1.160
11
1.303
Tabla 4. Segunda curva de calibración
Soluciones
Absorbancia
1
-0.000
2
0.010
3
0.122
4
0.179
5
0.397
6
0.635
7
0.738
8
0.925
9
1.131
10
1.240
11
1.406
Con base a los resultados obtenidos podemos concluir que se cumplió con los objetivos planteados en esta práctica. Se realizó correctamente la técnica de espectrofotometría UV- vis que permitió realizar las curvas de calibración para azúcares reductores y proteínas correctamente.
Los modelos matemáticos que definen a estas curvas de calibración fueron у= 0.666х + 0.1075 para Azúcares reductores, y у = 0.0021х + 0.0035 para Proteínas. Ambos con una R cuadrada superior a 0.98 lo que nos da la confianza para utilizarlas en futuros trabajos con estas sustancias.
Discusión
Referencias
Tabla 3. Primera curva de calibración
| |
Soluciones
|
Absorbancia
|
1
|
-0.001
|
2
|
0.001
|
3
|
0.073
|
4
|
0.195
|
5
|
0.366
|
6
|
0.534
|
7
|
0.685
|
8
|
0.834
|
9
|
1.032
|
10
|
1.160
|
11
|
1.303
|
Tabla 4. Segunda curva de calibración
| |
Soluciones
|
Absorbancia
|
1
|
-0.000
|
2
|
0.010
|
3
|
0.122
|
4
|
0.179
|
5
|
0.397
|
6
|
0.635
|
7
|
0.738
|
8
|
0.925
|
9
|
1.131
|
10
|
1.240
|
11
|
1.406
|
Con base a los resultados obtenidos podemos concluir que se cumplió con los objetivos planteados en esta práctica. Se realizó correctamente la técnica de espectrofotometría UV- vis que permitió realizar las curvas de calibración para azúcares reductores y proteínas correctamente.
Los modelos matemáticos que definen a estas curvas de calibración fueron у= 0.666х + 0.1075 para Azúcares reductores, y у = 0.0021х + 0.0035 para Proteínas. Ambos con una R cuadrada superior a 0.98 lo que nos da la confianza para utilizarlas en futuros trabajos con estas sustancias.
Discusión
Referencias
Castro, M., Litter, M., Wong, M., & Mori, V. (2009). Metodologías analíticas para la determinación y especiación de arsénico en aguas y suelos (Primera ed.). Argentina: CYTED.
Delgado, M. (2009). Utilización de Espectroscopía de Reflectancia en el Infrarrojo Cercano (NIRS), para la predicción del contenido de almidón en 18 variedades de papa nativa (Solanum tuberosum – ssp. tuberosum). Universidad Austral de Chile. Escuela de Ingeniería en Alimentos, pp. 23, 24.
Dosa, M., & Villanueva, M. (2008). Introducción a la metrología química. Curvas de calibración en los métodos analíticos. Extraído de: http://depa.fquim.unam.mx/amyd/archivero/CURVASDECALIBRACION_23498.pdf
Fanglian He. (2011). Bradford Protein Assay. 16.02.17, de Bio-Protocol Sitio web: http://www.bio-protocol.org/e45
Fuentes, X., Castiñeiras, J., & Queraltó, J. (1998). Bioquímica clínica y patología molecular (Segunda ed.). Barcelona: Reverté.
García, M., Colom, M., & Jaramillo, J. (2003). Manual del auxiliar de laboratorio. Temario general (Segunda ed.). Sevilla: MAD, S.L.
Guzmán, P., García, G. and Larios, E. (2013). Determinación de azúcares reductores método DNS. Fundación Universidad de América: Campus de los Cerros, p.1. Disponible en: http://www.academia.edu/4403544/DETERMINACION_DE_AZUCARES_REDUCTORES_METODO_DNS [Recuperado el 16 de febrero de 2017].
Ian M. Rosenberg. (2013). Protein Analysis and Purification: Benchtop Techniques. United States of America: Springer Science & Business Media.
John M. Walker. (2008). The Protein Protocols Handbook. United States of America: Springer Science & Business Media.
McKee, T., McKee, J., Araiza Martínez, M. y Hurtado Chong, A. (2014). Bioquímica. Las bases moleculares de la vida. 4° ed. Madrid: McGraw-Hill Interamericana, p.235.
Miller, G. (1959). Use of Dinitrosalicylic Acid Reagent for Determination of Reducing Sugar. Analytical Chemistry, 31(3), pp.426-428.
NPTEL. (2014). Determination of Protein Concentration by Bradford Method. 16.02.17, de NPTEL Sitio web: http://nptel.ac.in/courses/102103047/5
Ospina,M. (2008). Montaje de curvas de calibración para análisis de gomas, fosfatos, sílice, azúcar y sulfitos por espectrofotometría UV-Visible. Universidad tecnológica de pereira. Extraído de: http://repositorio.utp.edu.co/dspace/bitstream/handle/11059/1106/6641227O83.pdf?sequence=1
UAEMex (2012). Estadística. Correlación Lineal. [PDF] Estado de México: Facultad de Contaduría y Administración, p.5. Disponible en: http://www.seduca2.uaemex.mx/ckfinder/uploads/files/u3tema_1_coeficiente.pdf [Recuperado el 18 de febrero de 2017].
UNAM, (2012). Carbohidratos. [PDF] México, D.F.: AMYD, p.3. Disponible en:: http://depa.fquim.unam.mx/amyd/archivero/CARBOHIDRATOS_21119.pdf [Recuperado el 16 de febrero de 2017].
Reyes, J. y Quiroa, W. (2012). Curvas de Calibración y Método Estadístico de Mínimos Cuadrados. [PDF] USAC: Departamento de Fisicoquímica, p.2. Disponible en: https://sisoyyomismo.files.wordpress.com/2012/02/practica-2-instrumental-qb.pdf [Recuperado el 18 de febrero de 2017].






CASINOS in Miami casino in Miami | JTM Hub
ResponderEliminarMiami's Miami Casino is one 천안 출장안마 of four 광양 출장샵 casinos owned by Vici Properties. 부산광역 출장샵 Vici Properties 용인 출장마사지 owns the Wynn 순천 출장마사지 Hotel and Casino.